Résumé des travaux de recherche
Fanta Fall
Docteur en science de la vie et de la santé
Mes travaux de recherche s’inscrivent à l’interface de la chimie et de la biologie. Après une maîtrise en ingénierie chimique et biologique, j’ai préparé une thèse de sciences en sciences de la vie et en santé humaine. Mes travaux consistent à utiliser la chimie analytique, et plus particulièrement la spectrométrie de masse, dans des problématiques de santé humaine. La spectrométrie de masse et ses nombreuses applications en métabolomique et protéomique restent aujourd’hui au cœur de mes préoccupations scientifiques, notamment lorsqu’elles sont appliquées à la recherche en santé.
Travaux antérieurs au doctorat
Développements méthodologiques par spectrométrie de masse pour l’étude statique et dynamique du métabolisme des lipoprotéines.
Stages de Master 1 (6 mois, 2015) et Master 2 (6 mois, 2016) réalisés sous la direction du Professeur Michel Krempf et du Docteur Mikael Croyal. INRA UMR 1280, Physiopathologie des adaptations nutritionnelles.
Contexte biologique – Les apolipoprotéines constituent une famille de protéines circulantes solubles, assurant la structuration et la régulation des lipoprotéines (VLDL, IDL, LDL, HDL) en charge du transport des lipides dans la circulation sanguine (Figure 1). Les altérations du métabolisme des lipoprotéines exposent à un risque élevé de maladies métaboliques et cardiovasculaires1. La modulation des concentrations des apolipoprotéines peut être étudiée par l’analyse des flux métaboliques, aujourd’hui réalisée via l’utilisation de précurseurs enrichis en isotopes stables (ex : d3-Leucine). Les traceurs isotopiques sont perfusés aux patients et des prélèvements sanguins sont ensuite réalisés régulièrement. L’incorporation du traceur dans les apolipoprotéines est mesurée, et les taux de production et de catabolisme sont déduits des courbes cinétiques générées (Figure 2)2.
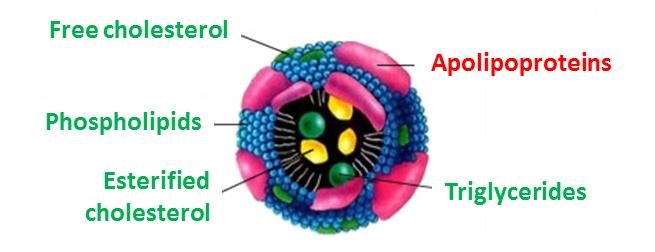
Figure 1 : Représentation schématique d’une lipoprotéine
Problématique initiale et hypothèse de recherche – L’analyse des flux protéiques nécessitait traditionnellement le recours à plusieurs techniques d’analyse souvent complexes (ELISA, électrophorèse/GC-MS) et donc limitées aux apolipoprotéines abondantes comme l’apoB100 de la voie VLDL → IDL → LDL et l’apoA-I des HDL. Récemment, des oligonucléotides antisens visant à réduire les niveaux d’apolipoprotéine C-III (apoC-III) ont été proposés pour combattre les troubles lipidiques de patients à haut risque cardiovasculaire3. L’apoC-III est en effet un facteur important de dyslipidémie et de risque de maladie cardiovasculaire, notamment dans le cadre de l’insulino-résistance et du diabète de type 24. Chez l’Homme, des taux plasmatiques élevés d’apoC-III sont associés à une hypertriglycéridémie et à une augmentation du nombre de particules de LDL athérogènes, petites et denses. Cependant, l’apoC-III est une apolipoprotéine de concentration moyenne, majoritairement portée par les VLDL et les HDL et dont les flux cinétiques étaient difficiles à étudier par les méthodes traditionnelles. Nous avons donc cherché à développer une nouvelle approche par spectrométrie de masse afin d’étudier de façon plus fine le métabolisme de l’apoC-III.
Approches méthodologiques mises en œuvre –Les méthodes analytiques basées sur la protéolyse enzymatique et l’analyse ciblée de peptides signatures par LC-MS/MS pourrait permettre la simplification des protocoles cinétiques utilisant des traceurs isotopiques5. Nous avons choisi de développer cette approche pour l’analyse cinétique et quantitative de l’apoC-III (Figure 2).
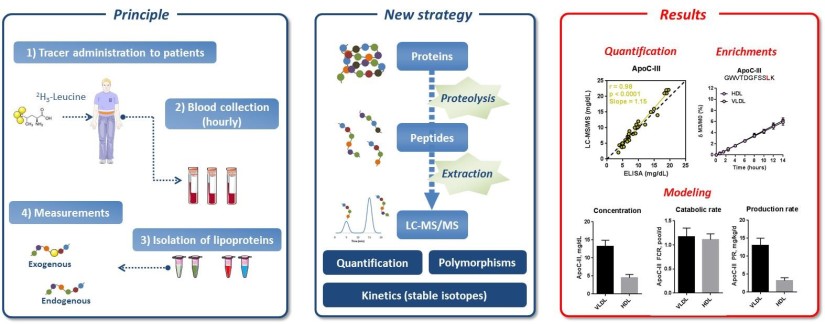
Figure 2 : Worklow général de l’approche analytique développée
Principaux résultats et nouvelles hypothèses – La LC-MS/MS a été comparée à l’ELISA pour l’aspect quantitatif et aux données de la littérature pour la mesure cinétique des enrichissements en d3-Leucine (approche classique par électrophorèse/GC-MS). Nous avons d’abord montré que la LC-MS/MS permettait l’obtention de données fiables et répétables (Figure 2). Par ailleurs, les paramètres cinétiques calculés à partir des données LC-MS/MS étaient conformes à ceux de la littérature obtenus via les méthodes conventionnelles. Comme l’approche analytique développée présentait l’avantage de pouvoir analyser plusieurs protéines en simultanée, nous avons amélioré notre approche LC-MS/MS pour le criblage cinétique et quantitatif de six apolipoprotéines majeures : apoA-I, apoA-II, apoB100, apoC-II, apoC-III et apoE. A nouveau, nous avons montré que la LC-MS/MS permettait l’obtention de données fiables en un temps beaucoup plus court, les six apolipoprotéines étant analysées simultanément alors qu’elles étaient étudiées séparément avec les méthodes antérieures. Par ailleurs, les paramètres cinétiques calculés à partir des données LC-MS/MS se sont encore avérés conformes à ceux de la littérature (méthodes conventionnelles). Ces travaux ont ainsi fait l’objet d’une publication dans Journal of Lipid Research, dans laquelle je suis deuxième auteur6. Nous avons par la suite exploré les contours de notre approche LC-MS/MS en affinant notre mode opératoire pour la quantification de protéines de plus faibles abondances telles que PCSK9, un inhibiteur naturel du récepteur aux LDL. Ces travaux ont fait l’objet d’une publication dans Journal of Chromatography B, dans laquelle je suis co-premier auteur7.
Contribution à l’évolution des connaissances – A la suite de mes travaux de stage, la méthode LC-MS/MS a encore été améliorée. Elle permet aujourd’hui l’analyse simultanée de dix-huit apolipoprotéines [(a), A-I, A-II, A-IV, A-V, B48, B100, C-I, C-II, C-III, C-IV, D, E, F, H, J, L1 et M] en une injection et à partir d’une faible prise d’essai (< 50 µL). Cette méthode est également capable de mesurer les enrichissements en d3-leucine dans des protocoles cinétiques et de quantifier certains polymorphismes [taille de l’apo(a), apoE2/E3/E4, apoL1 G0-G1-G2]. Cette méthode fait aujourd’hui l’objet d’une validation rigoureuse selon les référentiels cliniques et analytiques traditionnels et à laquelle je participe (publication en cours de soumission). De façon plus générale, mes travaux ont permis d’accroitre le niveau des connaissances du métabolisme des apolipoprotéines via diverses applications cliniques et physiopathologiques et m’ont permis d’être co-auteur dans trois autres publications scientifiques8–10.
Travaux de thèse
Etude métabolomique de la polarisation des macrophages pulmonaires humains
Sous la direction du Docteur Stanislas Grassin-Delyle et soutenue le 7 octobre 2019. INSERM U1173, Infection et inflammation, UFR santé-Simone Veil.
Contexte biologique – Les macrophages pulmonaires sont des cellules immunitaires impliquées dans la physiopathologie des maladies pulmonaires inflammatoires telles que l’asthme. Ils sont essentiels à la défense contre les agents pathogènes qui colonisent les voies respiratoires, et peuvent subir une polarisation vers deux phénotypes appelés M1 et M2. Le phénotype M1 est induit par les agonistes des Toll-like Récepteurs (TLR) et promeut la réponse inflammatoire, tandis que le phénotype M2 est induit par les cytokines Th2 canoniques (IL-4 et IL-13) et exerce principalement des fonctions immuno-régulatrices. Cette différenciation se traduit par des modifications du phénotype (expression de protéines membranaires, production de cytokines) et des fonctions cellulaires (phagocytose). La polarisation a aussi un impact sur le métabolisme et la production de médiateurs intracellulaires . A titre d’exemple, les oxystérols ont été récemment décrits comme fortement impliqués dans la réponse inflammatoire des macrophages aux agonistes des TLR. Une augmentation de la population de macrophages M2 a été décrite dans les maladies telles que l’asthme, la bronchopneumopathie chronique obstructive (BPCO) ou le cancer pulmonaire. La polarisation des macrophages fait l’objet de recherches intensives, mais les modifications du métabolisme cellulaire pendant la polarisation des macrophages pulmonaires humains n’avaient encore jamais été explorée .
Problématique initiale et hypothèse de recherche – L’étude de l’immuno-métabolisme des différents métabolites présents dans les macrophages est un domaine en plein essor. En revanche, aucun travail publié n’avait caractérisé les altérations cellulaires liées à la polarisation M1/M2 des macrophages pulmonaires, que ce soit au niveau du métabolome au sens large ou plus particulièrement au niveau de la voie des oxystérols. Les études publiées sur la polarisation des macrophages ont portées principalement sur des modèles de macrophages dérivés de monocytes (MDM) ou sur des modèles murins ne se comportant pas totalement comme les cellules primaires humaines. L’étude du métabolisme cellulaire humain via des approches métabolomiques pourrait amener à une exploration plus approfondie du métabolisme cellulaire dans de nombreux contextes. Au cours de mes travaux de thèse, j’ai développé des méthodes d’analyses métabolomiques ciblées et non-ciblées par spectrométrie de masse, dans le but de confirmer les différences observées au niveau transcriptomique suivant l’état de polarisation des macrophages pulmonaires humains.
Approches méthodologiques mises en œuvre – Pour analyser le métabolome humain et couvrir un large spectre de molécules, des méthodes métabolomiques non-ciblées par LC-HRMS et par GC-MS ont été employées. La première partie de mes travaux a consisté à développer une méthode optimale de préparation d’échantillon et d’acquisition des données non-ciblées, ainsi que des méthodes bio-informatiques de traitement de données. Dans cette partie, différentes méthodes de chromatographie ont été testées avec plusieurs types de colonnes chromatographiques. La spectrométrie de masse a aussi été optimisée en testant plusieurs paramètres de source et d’acquisition. Ce travail a été réalisé en étroite collaboration avec le CEA, (Etienne Thevenot et Natacha Lenuzza, MetaboHub) et a été appliqué à un modèle in vitro de polarisation M1/M2 avec des macrophages pulmonaires humains. Notre but était de mieux caractériser les mécanismes de l’inflammation pulmonaire et de sa résolution.
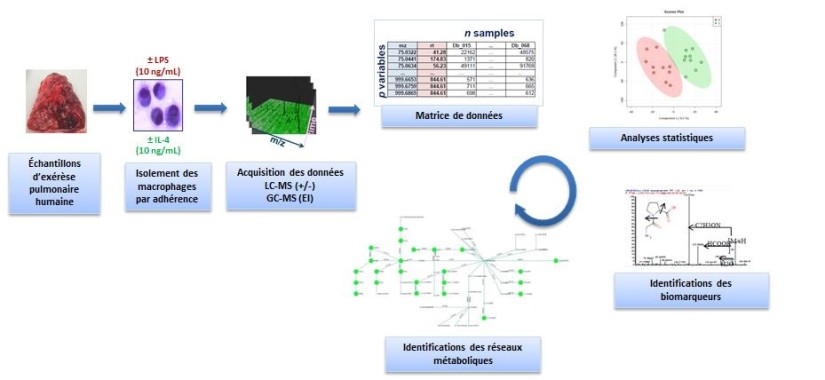
Principaux résultats et nouvelles hypothèses – Nous nous sommes appliqués à améliorer l’ensemble du processus analytique, de la préparation optimale des échantillons au traitement des données générées. Une nouvelle méthode d’acquisition originale a été développée et validée par segmentation de masse (m/z : 60-300, 300-600 et 600-900). Nos travaux ont permis d’améliorer considérablement l’intensité et la résolution du signal en favorisant la détection de métabolites et en minimisant les interférences (le bruit de fond), en comparaison avec le mode « full scan » à un seul segment de masse utilisé traditionnellement en métabolomique. Nous avons également confirmé l’intérêt de combiner les méthodes d’analyses non-ciblées aux méthodes ciblées pour la compréhension de systèmes métaboliques complexes. Les analyses non-ciblées permettent en effet l’identification de nouveaux biomarqueurs qui seront explorés quantitativement par les analyses ciblées. De la même manière, nous avons montré l’intérêt de combiner plusieurs types de colonne chromatographique et de systèmes analytiques pour une meilleure couverture du métabolome.
Après application aux macrophages pulmonaires humains, nous avons pu mettre en évidence quatre voies métaboliques :
- La voie de l’acide arachidonique
- La voie des kynurénines
- Le cycle de Krebs
- Les oxystérols
L’étude métabolomique a montré une régulation différentielle des métabolites appartenant à la voie des kynurénines, de l’acide arachidonique et du cycle de Krebs. Tous les métabolites identifiés ont pour point commun d’intervenir dans l’entretien et la régulation de la réaction inflammatoire. Pour certaines de ces voies, nous avons développés des méthodes d’analyses ciblées pour quantifier un maximum de métabolites impliqués. Cette étude nous a permis d’identifier les différents profils de macrophages pulmonaires polarisés M1 et M2 et a fait l’objet d’une publication dans Journal of chromatography B, dans laquelle je suis première auteur et une en soumission dans PlosOne.
Contribution à l’évolution des connaissances – La méthode d’acquisition des données par LC-HRMS a été utilisée pour les autres études métabolomiques du laboratoire. Ces travaux ont également permis de mettre en évidence certaines voies métaboliques majeures dans les pathologies pulmonaires. Nos travaux ont permis de développer des méthodes d’analyses métabolomiques intéressantes sur des échantillons de culture cellulaire. La méthodologie pourra ensuite être appliquée aux tissus pulmonaires de patients asthmatiques, BPCO ou souffrant de mucoviscidose dans le but d’identifier de nouvelles cibles moléculaires impliquées dans la régulation de la polarisation au cours de ces pathologies. Ceci permettra d’évaluer différents traitements administrés aux patients sur l’inflammation pulmonaire et sa résolution.
Travaux postdoctoraux
Étude métabolomique des agents antiparasitaires naturels
Pharmacognosy Research Group, Louvain Drug Research Institute (LDRI), Université catholique de Louvain, Brussels, Belgium.
Les laboratoires de pharmacognosie de l’ULiège et de l’UCL ont identifié ces dernières années plusieurs produits naturels possédant des propriétés antipaludiques et antitrypanosomiales prometteuses. Le mode d’action de ces composés n’est pas ou pas complètement connu, même si pour certains d’entre eux, des cibles originales semblent être impliquées. Les objectifs de ce projet sont d’appliquer la métabolomique MS et RMN à l’identification des voies métaboliques et / ou biochimiques modifiées par ces dérivés antiparasitaires naturels prometteurs, chez Trypanosoma brucei et Plasmodium falciparum. Cela permettra de détecter les changements induits dans le métabolisme cellulaire, de déterminer si ces molécules possèdent des modes d’action spécifiques et d’identifier de nouvelles cibles thérapeutiques potentielles qui pourraient être étudiées dans d’autres modèles. La découverte de nouvelles cibles putatives est clairement une priorité dans le contexte de la résistance des parasites aux médicaments actuellement utilisés. »
Références
- Dominiczak MH, Caslake MJ. Apolipoproteins: metabolic role and clinical biochemistry applications. Ann Clin Biochem. 2011;48:498–515.
- Barrett PHR, Chan DC, Watts GF. Thematic review series: patient-oriented research. Design and analysis of lipoprotein tracer kinetics studies in humans. J Lipid Res. 2006;47:1607–1619.
- Tsimikas S. RNA-targeted therapeutics for lipid disorders. Curr Opin Lipidol. 2018;29:459–466.
- Koska J, Yassine H, Trenchevska O, Sinari S, Schwenke DC, Yen FT, Billheimer D, Nelson RW, Nedelkov D, Reaven PD. Disialylated apolipoprotein C-III proteoform is associated with improved lipids in prediabetes and type 2 diabetes. J Lipid Res. 2016;57:894–905.
- van den Broek I, Sobhani K, Van Eyk JE. Advances in quantifying apolipoproteins using LC-MS/MS technology: implications for the clinic. Expert Rev Proteomics. 2017;14:869–880.
- Croyal M, Fall F, Ferchaud-Roucher V, Chétiveaux M, Zaïr Y, Ouguerram K, Krempf M, Nobécourt E. Multiplexed peptide analysis for kinetic measurements of major human apolipoproteins by LC/MS/MS. J Lipid Res. 2016;57:509–515.
- Croyal M, Fall F, Krempf M, Thédrez A, Ouguerram K, Ferchaud-Roucher V, Aguesse A, Billon-Crossouard S, Mata P, Alonso R, Lambert G, Nobécourt E. Plasma PCSK9 measurement by liquid chromatography-Tandem mass spectrometry and comparison with conventional ELISA. J Chromatogr B Analyt Technol Biomed Life Sci. 2017;1044–1045:24–29.
- Croyal M, Billon-Crossouard S, Goulitquer S, Aguesse A, León L, Fall F, Chétiveaux M, Moyon T, Blanchard V, Ouguerram K, Lambert G, Nobécourt E, Krempf M. Stable Isotope Kinetic Study of ApoM (Apolipoprotein M). Arterioscler Thromb Vasc Biol. 2018;38:255–261.
- Croyal M, Kaabia Z, León L, Ramin-Mangata S, Baty T, Fall F, Billon-Crossouard S, Aguesse A, Hollstein T, Sullivan DR, Nobecourt E, Lambert G, Krempf M. Fenofibrate decreases plasma ceramide in type 2 diabetes patients: A novel marker of CVD? Diabetes Metab. 2018;44:143–149.
- Kaabia Z, Poirier J, Moughaizel M, Aguesse A, Billon-Crossouard S, Fall F, Durand M, Dagher E, Krempf M, Croyal M. Plasma lipidomic analysis reveals strong similarities between lipid fingerprints in human, hamster and mouse compared to other animal species. Sci Rep. 2018;8:15893.
- Shapouri-Moghaddam A, Mohammadian S, Vazini H, Taghadosi M, Esmaeili S-A, Mardani F, Seifi B, Mohammadi A, Afshari JT, Sahebkar A. Macrophage plasticity, polarization, and function in health and disease. J Cell Physiol. 2018;233:6425–6440.
- Biswas SK, Mantovani A. Orchestration of metabolism by macrophages. Cell Metab. 2012;15:432–437.
- Victoni T, Salvator H, Abrial C, Brollo M, Porto LCS, Lagente V, Naline E, Grassin-Delyle S, Devillier P. Human lung and monocyte-derived macrophages differ with regard to the effects of β2-adrenoceptor agonists on cytokine release. Respir Res [Internet]. 2017 [cited 2019 Jul 16];18. Available from: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC5480184/
- Groot-Kormelink PJ, Fawcett L, Wright PD, Gosling M, Kent TC. Quantitative GPCR and ion channel transcriptomics in primary alveolar macrophages and macrophage surrogates. BMC Immunol. 2012;13:57.
- Fall F, Lenuzza N, Lamy E, Brollo M, Naline E, Devillier P, Thévenot E, Grassin-Delyle S. A split-range acquisition method for the non-targeted metabolomic profiling of human plasma with hydrophilic interaction chromatography – high-resolution mass spectrometry. J Chromatogr B. 2019;1128:121780.
